CRISPR/cas9为基础的基因筛选为生物学问题的探究提供了新的工具好思路,通过简单的改变sgRNA,我们可以将cas9引导到基因组的不同位置。但是sgRNA存在着不同的on-target活性和off-target问题。
那么如何根据筛选得到结果,预先确定一些有效的sgRNA?
今天带大家一起走进David E Root课题组的工作,了解统计分析在sgRNA确定上的神奇与伟大。很多研究目前已经确定Cas9 脱靶的活性同时依赖于sgRNA的序列和不同的实验条件。这些研究为初步了解脱靶问题提供了一定的研究基础,然而关于sgRNA脱靶的主要定量模型并未得到解析。
这里David E root课题组则通过基于前人发表的Human和mouse screen的结果,对于sgRNA进行的统计分析,提出了sgRNA活性和脱靶性的预测模型。
首先我们来看看2014年该课题组通过针对于不同基因设计的密铺sgRNA,进而评价sgRNA活性的工作。

为了发现影响sgRNA效率活性的序列特征和target区域特征,科学家们的策略是通过针对于细胞表面marker,进行sgRNA的设计,通过密铺设计所有可能的sgRNA,科学家们通过一个细胞递送一个sgRNA,然后通过FACS分离biallelic敲除细胞,然后通过测序,获得最高活性的sgRNA。
首先科学家们在EL4细胞系,老鼠胸腺细胞系进行的探究。经过9天的转导,对于内源性表达的Thy1,H2-K,Cd45, Cd43, Cd28,Cd5进行分选富集敲除细胞。在人的细胞系中则选取了MOLM13,NB4和TF1通过转导Cas9进而通过筛选富集活性sgRNA。
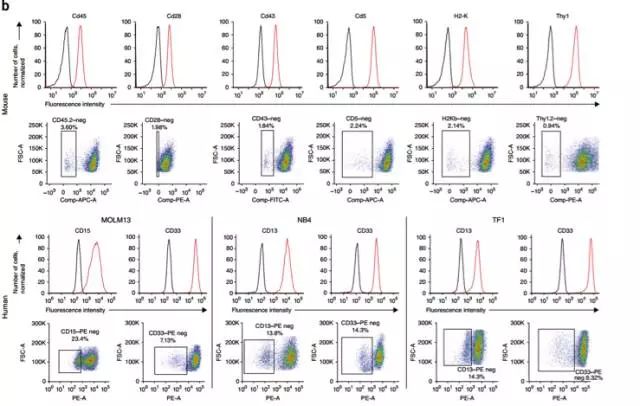
接下来,科学家们对所有的sgRNA在基因组的位置进行了注释,如下图,我们可以拿到在靠近前面extron位置的sgRNA活性会更高。
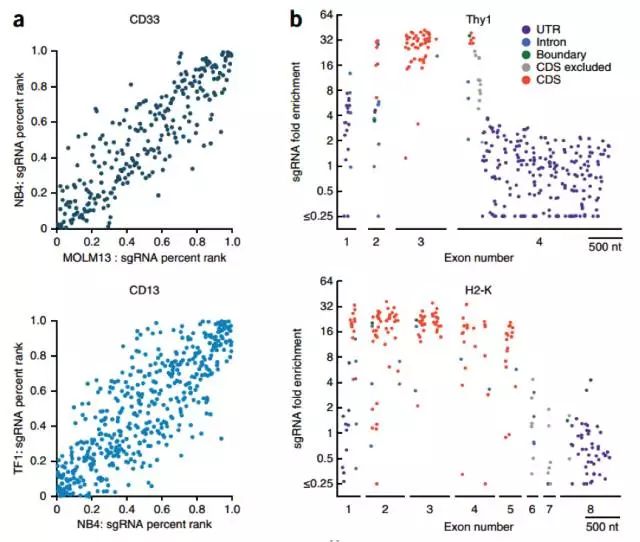
一些exon区域的sgRNA并未有活性,因此暗示这些exon并未表达在相应的细胞系中。同时在CD15的N端活性则不是很高,很有可能由于不同的局部表观遗传学修饰的影响。在获得sgRNA位置特征后,科学家们的目光转移到CDS区,期望能从中获得sgRNA序列特征。
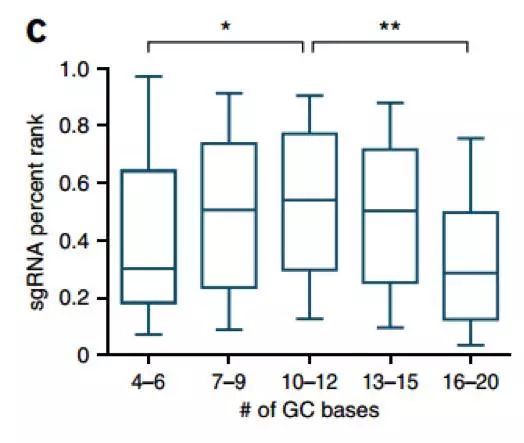
如以前发现,过高或过低的GC含量都会导致活性的降低。
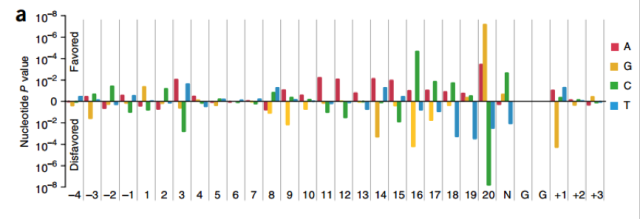
科学家们发现,在sgRNA20号位置的sgRNA更倾向于G而排斥C,在16位置的sgRNA则倾向于C排斥G,在sgRNA的中间区域存在A活性更高。不仅如此,科学家还发现针对于sgRNA的PAM区域,则CGGT的活性最高。针对于以上可提出来的准则,科学家们,则提供了sgRNA的活性的预测。
网址:
http://www.broadinstitute.org/rnai/public/analysis-tools/ sgrna-design

